
新学術領域研究「ノンコーディング RNA ネオタクソノミ」の公式ブログです。コメントはどなたでも歓迎します。
パラスペックル研究の生みの母、Archa FoxさんとCharlie Bondさん企画のプチシンポジウムに参加するため、オーストラリアはパース、University of Western Australiaに行ってまいりました。
ENCODEのshRNA-RNAseqはこりゃ便利だということでここのところENCODEコンソーシアムから頼まれたわけでもないのにウルトラ超絶宣伝していますが、このデータをよくよく見てみると、実験群ごとにだいぶ様子が違うのに気付きます。例えば、下の図はとあるふたつのコントロール実験(control shRNAでノックダウンした実験)におけるACTBのリードの張り付き方を見たものですが、、、
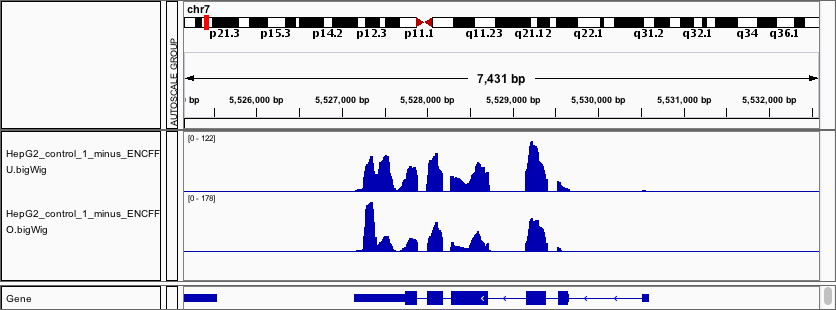
リードの張り付き方のパターンがえらく違います。
恥ずかしながら、僕自身、ウェットの研究者がピンセットとピペットマンだけ握っていれば良い時代はとうの昔に終わったことに気づいたのは、実はつい最近。今更データサイエンティストを目指すのもなんだしなあ、、、と半ば諦めてたところはあるのですが、ちょろっと触ってみるとこれが意外と楽しいんですね。HelloとShitとGreatだけの英会話みたいなレベルでも、意思疎通できるというのはそれだけで喜びを与えてくれるものです。それに加えてI love youだけ覚えれば人生の90%のミュニケーションは事足ります。というわけで、I love you R!
もう10年ほど前になりますが、Gomafuの核内局在因子やXistの染色体極在化因子を探そうとして僕らがとったアプローチは、RNA結合蛋白質に対してデザインしたカスタムsiRNAライブラリーを用いて片っ端からノックダウンしていって、その効果を調べて真打を探す、というものでした。HGWさんのたぐいまれなる強運もあり、このアプローチは大成功を収めたわけですが、ENCODEのshRNA-RNAseqのデータが使える今日だったら、実験を始める前からデータは手許にある!ということで、かなりの部分をショートカットすることができるわけです。というわけで、R初心者によるR講座第ウン段。今回は、自分の興味のある遺伝子の転写産物が、特定のRNA結合蛋白質をノックダウンした時にどのような変化をするのか、ということを可視化する方法を紹介します。まあ、そのうちUCSC Genome Browserでもすぐに見れるようになるんでしょうが、ちょっとだけ先を行こうということで、、、
「十年後の君しか見ない!」
研究者として自信がもてず、不安な日々を過ごしていた頃、運よくも“さきがけ研究”に選んでくださった時に、元熊本大学学長の江口吾郎先生に、最初に呼び出されてかけられた言葉がそれでした。
ベンチ屋がやるなんちゃってバイオインフォにとって欠かせないのは手軽に解析環境をインストールすることのできるMacOSとhomebrewなわけですが、homebrewでカバーできないツールも結構あります。ENCODEのデータがリリースされたのをきっかけにどりゃどりゃGomafuちゃんでも見てみようかと思った時に、一つあたり計算に数時間かかるCufflinksに多数のデータを突っ込むのは、ラボのMacProレベルでは小錦を自転車に乗せるようなものでとても無理。もっと手軽なカウントツールとしてHT-seqやらSubreadのfeatureCountやらあるわけですが、brew install HTseqやbrew install subreadではまだサポートされてないのでインストールできません。
タイトルの通りですが、Gene YeoさんやBrenton Graveleyさんが中心となって進めているENCODEのRNA結合蛋白質関連のプロジェクト論文がbioRxivにアップされました。
とにかくこれだけのデータが公共データベースで誰でも使えるようになっているというのがすごい!なんせ、200近いRNA結合蛋白質についてshRNAを用いたノックダウンのRNAseqのデータが手に入り、しかも半分ぐらいのものに関してはeCLIPのデータがセットになってるんですよ。これって一つのRNA結合蛋白質についてやるだけでも、普通に一つのラボが何年もかけてやる実験じゃないですか。多くのものについては免疫染色のデータもついてます。本当に一昔前なら信じられない状態です。今風に言うなら、やばい、やばい、やばい状態です。細胞はK562とHepG2と限られていますし、必ずしも自分の興味あるRNA結合蛋白質が全て含まれているわけではありませんが、特定のRNA結合蛋白質をノックダウンして発現量が変わるのかな、スプライシングのパターンが変わるのかな、転写開始位置やpoly-A付加位置が変わるのかな。これら全ての疑問は、ベンチで実験しなくても、データを解析するだけで答えが出る!!このデータを元にした再解析をするだけで、立派な論文が書けそうです。やってみようかな。いや、是非やってみましょうよ!
というわけで、eCLIPとshRNA-RNAseqのbamファイルを遺伝子の名前と細胞の名前つきでダウンロートできる簡単すぎる恥さらしでもちょっと役にたつかもしれないシェルスクリプト用意しました。ENCODEのサイトにも一括ダウンロードできるファイルがあるのですが、前記事でも触れた通り、Accession#しかファイル名がないので、直感的に使いにくいんですね。とりあえずダウンロードしておけば、好きな時にいくらでもデータにアクセスできます。そのうちUCSC Genome Browserにリンクがつくのでしょうが、それまで待てない!という感じですね。
しかし全部ダウンロードすると10TBぐらいにはなりそう。HDDをまず買いに行かなければ、、、
研究室は3回引越して、自宅は2回引越して海外にも住んで、研究室の人員も年々入れ替わって。そういう変化する日常の一方で、長年あまり変わらないものもあります。
アガサクリスティの「蒼ざめた馬」の冒頭シーン。一人の男をめぐって二人の女性が掴み合いの喧嘩をする強烈なシーンがありますが、そういうシーンはドラマではよくある展開ですが、現実世界ではほとんど見たことはありません。都市伝説みたいなものだと思っていたら、30年ほど前、某旧友をめぐって某女子校で似たようなシーンが展開されていたとかいう話をつい最近友人より聞きまして、そうか、そういう世界もあるんだなあと非モテ系の僕としてはちょっぴりというかだいぶ羨ましい、とかなんとか言っていると奥さんにキッと睨まれそうですが、プライベートではともかく、仕事で使っているモデルマウスが引っ張りだこというのは文句なしに嬉しいものです。そう。国内外からリクエスト来まくりの引っ張りだこのモテ男くん、Neat1 KOマウスの話です。昨年、今年と、このモテ男くんを使ったガン研究の論文がChris MarineさんとLaura Attardiさんのところから発表されました。これがややこしいことに一見すると正反対の結論。片や、Neat1がなくなるとガンになりにくい。片や、Neat1がなくなるとガンになりやすい。一体どっちなの?ということなのですが、両グループの共同研究者として、僕なりの理解をちょっとまとめてみようと思います。