頼みの綱のbiopapyrusさんのHPを頼りにインストールしようとしても、まずしょっぱなの
pip install numpy --userから、
-bash: pip: command not foundという非常なメッセージ。
これだからベンチ屋は、、、と、世の中のバイオインフォの専門家の悲嘆が聞こえてくるような気もしますが、いろいろなツールをマニュアル片手に入れようとしても、依存関係のあるパッケージのどれ一つが抜けてもエラーが出ますし、前のiMacではうまくいったのに今のMacProではうまくいかないとか落とし穴満載です(原因はえーと、、、察してください)。どこかに便利なやり方はないかと思い悩んでいたら、python系のツールはAnacondaを使えば一発ですよ、とラボの米田さん。持つべきものは良き同僚です。とりあえずAnacondaのインストールページに飛びます。最新版では使えないツールもあるということで、安定のPython2.7版をダウンロード。見慣れたMacOSのインストール画面に従って、Anacondaをインストール。Macintosh HDの直下においてしまって良いようです。Minicondaという簡易版もあるようですが、信じるものは救われる。Anacondaにしておく方が良いようです。プロトコールは省略しない。伝説だと思ってもFISHでアセチル化のステップを省かないようなものですね。(追記:biocondaとかminicondaとかだとPATHの設定等があってトーシロにはちょっとだけハードルが高くなるみたいです)
で、ターミナルを立ち上げて、Anacondaがインストールされているかを確認
conda list
ごちゃごちゃ管理しているパッケージの一覧が出て来ればオーケーです。もしも無反応ならパスを通します。
export PATH=~/anaconda/bin:"$PATH"
そこで、ツールをインストール
conda install -c bioconda htseq conda install -c bioconda subread
おーっ!!画面が流れ始めてなんかやってます。homebrewを彷彿させる、何もしてないのに仕事してる感が味わえるこの感覚。さて、実際に使えるかコマンドを打ってみます。試しにNOL12のeCLIPをカウントしてみます。
featureCounts -T 4 -O -a refseq.gtf -o result.txt HepG2_NOL12_1_ENCFF722JFH.bam
プログラムが走り始めて最初に出てくるのがこの表示。
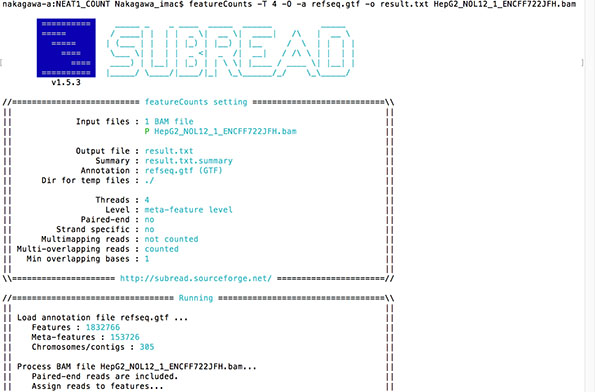
いにしえのマイコンBASICマガジンのプログラムに出てきそうなSUBREADの文字がたまりませんね。じゃあ計算してる間コーヒーでも飲んでくるかと腰を浮かせたところ、
|| Running time : 0.06 minutes ||
早っ!!数秒で終了!超高速参勤交代顔負けの超高速カウンターです。
ともあれ、python系のツールもanacondaに任せればhomebrew感覚ですぐに使えるというのは便利!いつまでたっても自力でインストールできないというのはそれはそれで問題なのかもしれませんが、ベンチ屋にとっての大きな味方です。ちなみにsubreadはRでも使えるみたいで、この場合、Rのコマンドラインで
source("https://bioconductor.org/biocLite.R")
biocLite(pkgs="Rsubread")
library ("Rsubread")
とすればもうこれでオーケー。Rが全て面倒を見て関連ツールをインストールしてくれます。何て簡単なんだ。結果が出てきたらそのままedgeRとかでの解析に入れるので、こっちの方が楽かもしれません。先ほどのコマンドラインと同じ命令をするとしたら
result=featureCounts(nthreads=4, allowMultiOverlap=TRUE, isGTFAnnotationFile=TRUE, annot.ext="refseq.gtf", files="HepG2_NOL12_1_ENCFF722JFH.bam")
ですね。この辺りのオプションのつけ方は慣れないと若干戸惑いますが、Subread/Rsubreadはマニュアルもとっても親切でわかりやすく、wet-friendlyなツールですね。
追記:
conda installに関してはボノさんのブログやImamachi-nさんのブログでも詳しく触れられていました。専門家の解説、参考になります!


 2020
2020