ところが、ものによってはなかなか簡単にいかないものがあります。最近気になっているのが、Arraystar社が出しているlncRNAに特化したマイクロアレイ(GPL15691)を使ったデーターセットで、例えばGSE85439という解析では、肝臓と脂肪と筋肉での遺伝子発現を、通常状態、24時間絶食、絶食後4時間、高脂肪食摂取後48時間後、高脂肪食摂取後12週後、それぞれn=4とか5のスケールで解析しています。なんじゃこれ!一体いくらかかったんだろうとNIHのCaoさんの懐具合を心配してしまいますが、それこそ余計なお世話というもので、使えるものはありがたく使わせていただく、ということで、「Analyze with GEO2R」のリンクをポチッとな。しっかしこれが分かりにくい。押してもらおうという覇気が全く感じられないリンクなので、キャプチャ画面つけておきます。
.jpg)
出てきた画面でDefine groupsでグループを定義し、Top250のボタンを押すと、グループ間で発現変動する遺伝子をすぐに調べられます。こちらも押してもらおうという覇気が全く感じられないリンクなので、一応キャプチャ画面をつけときます。
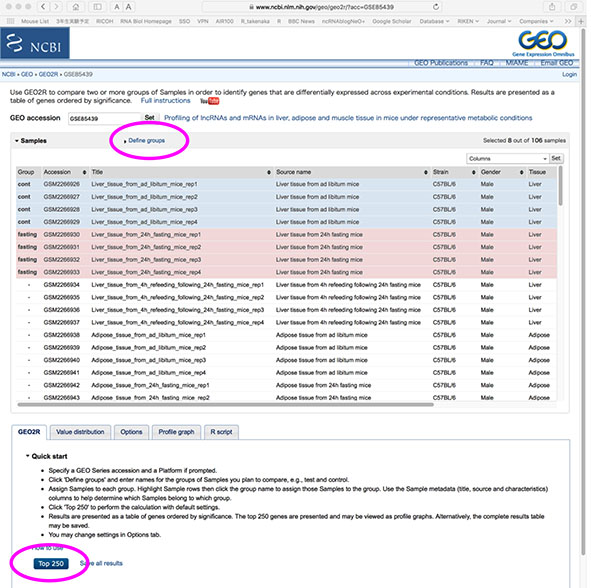
で、結果がこちら。
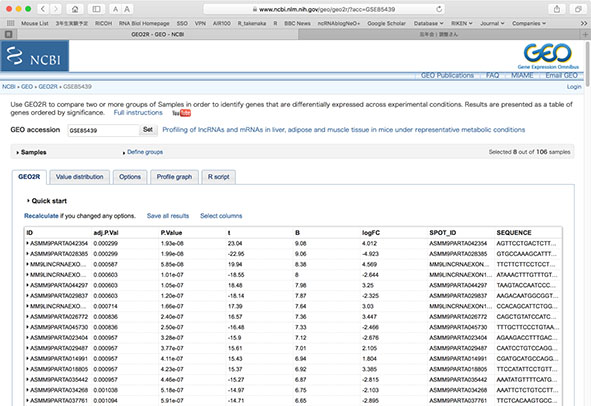
おー!素晴らしい。選んだサンプル間で発現変動がある遺伝子が一目瞭然!
ん?一目瞭然??
IDはあるけど遺伝子名がないではないか、、、
配列の情報があるのでそれをコピペしてBlatでもかければ遺伝子は判明するわけですが、自分の興味がある遺伝子、例えばGomafuとかGomafuとかGomafuとかの各サンプル間での発現変動を調べたい時だってあるわけです。そういう時は、Profile Graphというこれまたあまり覇気が感じられないタブの解析を選んでプローブIDを入れれば良いということはわかったのですが、そもそもそのプローブIDがわからない!
AffymetrixであればNetAffixやUCSC Geonme Browserから特定の遺伝子のprobe IDは簡単に引っ張ってこれますが、Arraystarは無料でデータを使おうとする輩にはIDは教えてあげないよん、という方針なのでしょうか、Google先生に聞いても、見つかりません。困った、困った、と思っていたところ、アレイの情報のページに行くと、プローブIDと配列の対応情報はすぐに見つかりました。
.jpg)
このアレイに関してはlncRNAに特化しているので、ncRNAblog+NEOを見られている方々も使う人もいるかもしれない、ということで、IDと配列だけのcsvファイル(Arraystar_probe_list.csv)を作って、このページの下のほうに置いておきました。(よくよく見るとこれも覇気の感じられないリンク、、、)
あとは、このファイルをエクセルで開いて、例えばC2のセルにGomafuの配列を入れて、
=IF(COUNTIF($C$2,"*"&B4&"*"),"Yes","No")
みたいにワイルドカード検索で部分一致したらYesというフラグを立てさせる式を書いて、下にががっとコピーしてて、後でソートすればオーケー。
なのですが、学生さんに「エクセルでなくRを使えRを!」と、自分で出来もしないことをいつも推奨している手前、Rのコンソールを使ってプローブIDの抽出もしてみました。
まずは、NCBIのNucleotideからGomafuの配列をとってきて、fasta形式のファイルをセーブします(Gomafu.fasta)。プローブリストのファイルと同じ作業ディレクトリに入れておきます。
次に、Rのコンソールを開いて、DNA配列操作関連の便利なツールのBiostringsをインストール
library(Biostrings)
後は、プローブとIDの対応表とGomafuの配列を読み込んで、部分一致したらその情報を出力させるだけ、
data<-read.csv("arraystar_probe_list.csv",stringsasfactors=f)
Gomafu<-readdnastringset("gomafu.fasta")
for (i in 1:59729) {
seq<-data[i,2]
if (grepl(seq,Gomafu)=="TRUE") {
print(data[i,],row.names=FALSE)
}
}
結果、
ID Probe
ASMM9PARTA044820 TTCATGCTTGTAGCTGCCTCTGTGTAAGATGCCATTTCAATATTAAAACCGACACACACT
ID Probe
ASMM9PARTA045976 GGTACAACACCAACCCACAAGGTTAGCAGCCCTTACCATTCCTCCACTTATGGGCCTTAT
ID Probe
ASMM9PARTA050182 TTACTTTGAGTTTTTCTGGGTCACACAAGAACCCACAATGCTCTAGGTGACAGAGGAGGA
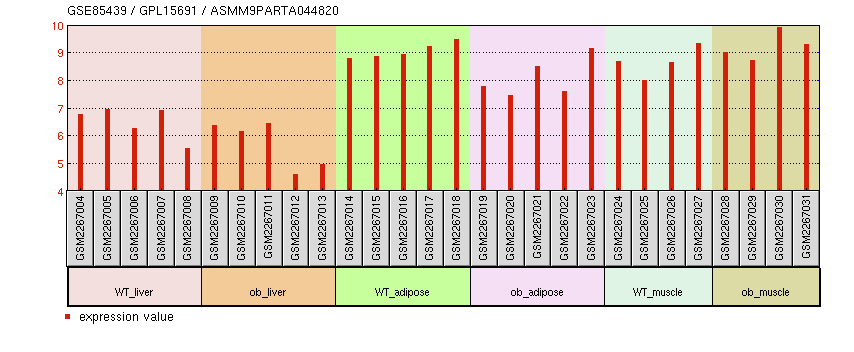
あとはこのIDをProfile GraphのIDに入れてポチっとな、するだけで、、、
まあ、あまり本業とは関係ないのですが、こんなデータがパッと出てきます。やたー!
(ハア、ハア、、、、息切れ寸前です)
インフォのプロからするとバカにされそうですが、なんちゃってコマンドラインはベンチ屋の気分転換には最高!ということでお許しを。ミニプレップでDNAが取れた!みたいなレベルですが、小さな喜びが日々の糧になるのも確かでして。。。
他にも、このArraystarのlncRNAを使ったデータとしては、
LncRNAs expression profiling in adipose tissues and during brown adipocyte differentiation
Long non-coding RNAs and microRNAs involved in integrated co-regulation of neuronal maturation
Long non-coding RNAs and mRNAs profiles in mouse liver development
などなど、があるようなので、暇な時にまたつらつらネットサーフィンならぬGEOサーフィンを楽しもうかと思います。


 2020
2020