おそらくですが、このshRNA-RNAseq。一度に全てのノックダウン実験を行うのは無理筋なので、何回かに分けて、時間も、もしかすると場所も異なる場所で行われていると思うのですが、その時の細胞の状態、RNA抽出からライブラリー作製のクオリティー、それらは完璧に揃わないのでしょう。ですので、そのノックダウン実験が、いつ、どのコントロール実験と一緒に行われたのかという情報は非常に重要な意味を持つと思われます。実際、個々のデータのサイトを見てみると、、
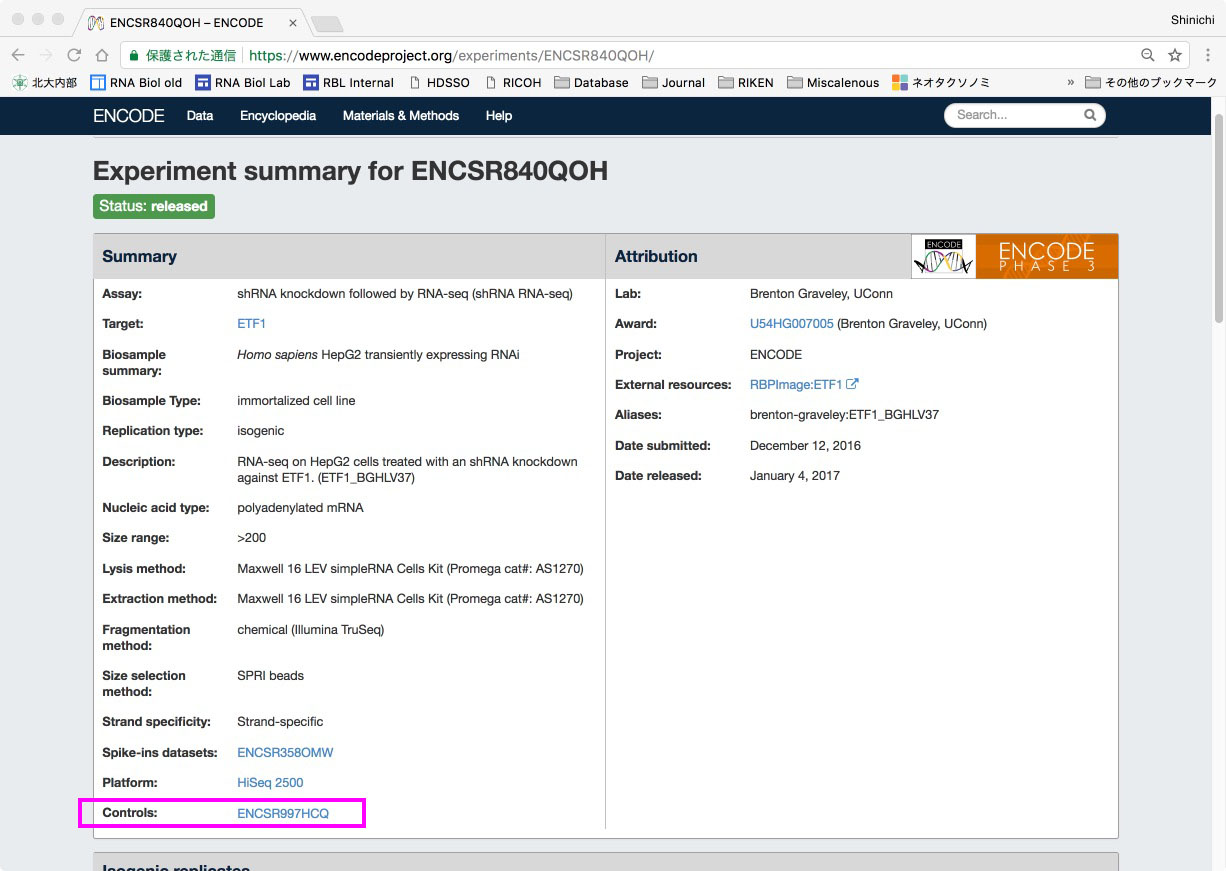
左下の方にcontrols: というところに、このノックダウン実験と同じ時に行ったコントロールノックダウン実験のIDが書いてあります。ところがこの情報は、上記のように個別の実験のサイトを訪れれば確かに書いてあるのですが、先日のこのブログでも紹介したENCODEが提供しているmetadata.txtには、どこを見ても入っていないんですよね。うーむ。それぐらい自分でなんとかしろということか、、、
というわけで、ENCODEの各実験のExperiment.accessionと対応するコントロール実験のExperiment.accessionを並べたファイルを作りました。えっちらおっちらリンクをコピペしてマニュアルで入力したものなので間違いがあるかもしれませんが、、、ENCODE提供のmetadata.tsvとこのmetadata_controlexp.txtのファイルをExperiment.accessionをキーにしてmergeしたものを用意しておけば、何かと役にたつかも?しれません。(注:なんかこの二つのファイルを単純にmergeコマンドで統合すると、all=FALSEにしても何故か行数が増えてしまうので、for文でくっつけてみました。もっと良いやり方がある、、、はず。)
meta=read.delim("metadata.tsv")
meta_cont=read.delim("metadata_controlexp.txt")
meta1=meta
num=nrow(meta_cont)
meta1$Control.experiment.accession=""
for (i in 1:num){
ID_exp=as.character(meta_cont$Experiment.accession[i])
ID_cont=as.character(meta_cont$Control.experiment.accession[i])
r1=grep(ID_exp,meta1$Experiment.accession)
meta1$Control.experiment.accession[r1]=ID_cont
}
write.table(meta1,file="metadata_plus.txt",quote=F,row.names = F,sep='\t')
しっかし、GAPDHの発現量一つとって見ても、結構コントロール実験ごとに違うんですよね。referenceとなる遺伝子の発現がバラツキまるときにどう考えるか。ちょっとTWでつぶやいたところ、速攻2分後に専門家の方からレプライがありました。SNS恐るべし。
FPKM of GAPDH in 24 control experiments in ENCODE shRNA-RNAseq data. Not adequate for the internal control gene? pic.twitter.com/5tPNvvM8PD
— Shinichi Nakagawa (@smoltblue) 2017年9月14日
That's known issue especially for human sample.
— Jun Seita (@jseita) 2017年9月14日
とはいえ、バラつくことが織り込み済みのGAPDHはさておき、GAPDHと並んでコントロール実験の双璧、ACTBでも、実際のところENCODEのデータを見る限り、GAPDHに比べればずっとマシですが、実験ごとに結構ばらつくようです。だからこそ、同じバッチでやったコントロール実験が大事なのだと思いますし、逆に言えば、これだけ標準化に気を配っているENCODEデータのACTBですらこれだけの振れ幅がありうる、と言うことは、肝に据えておいた方が良いことなのかな、と思います。
ACTB seems to be fairly constant, although still be quite variable. Perhaps within a range of natural variation. pic.twitter.com/GTqbOe1aRk
— Shinichi Nakagawa (@smoltblue) 2017年9月14日
ですので、揃いすぎているエラーバーの方が、
「ちょっと待て このエラーバー ありえない」
みたいな。むしろある程度バラつくことこそが真実、みたいな印象がありますね。


 2020
2020