というところまででした。で、何が起きたかと言いますと、予想されたサイズにはバンドが出ず、90ntのところに強いバンドが出たわけです。すわ、こりゃレトロトランスポゾンへのクロス再びか!とおもったのですが、repeat maskerでレトロトランスポゾンが検出されなかったプローブです。おかしいなあ、おかしいなあ、というわけで、とりあえずプローブを細かくちぎって、どの領域が90ntのRNAを検出するか絞り込みを行っていきました。当時はゲノムプロジェクトはとっくに終わっていましたが、なにか見逃されている配列があるのかもしれない、そのRNAは特殊な修飾がかかっていて逆転写されないとかあるのかもしれない、と妄想はどんどん膨らんでいって、ちょうどRNAフロンティアミーティングの特別講演をお願いした縁でお知り合いになった鈴木勉さんに、絞り込んだプローブを用いてRNA精製>Mass解析というとてつもなく手のかかるお仕事を共同研究でお願いする一方(実際の手を動かしてくださった宮内さん、お手数をおかけしました、、、)、いろいろなデータベースでしつこく検索やりなおしというのをちらほらやっていたところ、ん?NCBIのblastで、AK037328の一部の配列が灰色になっていることに気がつきました。これって、もしかしてリピート配列??そこでまたいろいろデータベースを当てまくったら、lncRNAdbだったかなんだったか、4.5SHという配列に似てますよ、と返してくるではないですか。そこでその配列をよく見てみると、
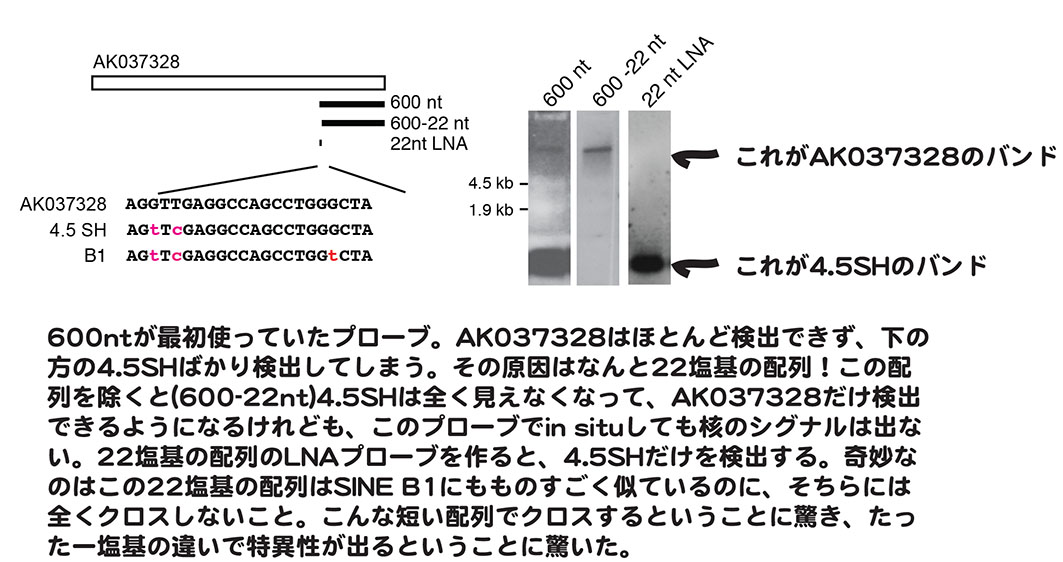
はああ、、、22nt中20ntしか一緒じゃないけど(しかも2ntミスマッチ入っているし)、たしかに相同性はある。驚くべきことに、RNaseA処理を省いたin situ hybridizationでは、こんなに短い配列の相同性も拾ってきてしまうのですね。いやあ、、、と、ため息しかつけなかったのですが、ちょうどこれに気づいた頃、鈴木研の超絶MASS解析の結果も上がってきて、こりゃどうやら4.5SHですねえと。インフォでカタがつく実験だったのにめちゃくちゃ手間のかかる生化学的実験で大変お手数をおかけしてしまって本当にバカバカバカと自分をポカポカポカしたくなりましたが、一応、全ての謎が解決してスッキリしたという意味では間違いなく進歩です。というわけで、気を取り直して(何回気を取り直しているんだか、、、)ここはひとつ、流れ弾みたいだけど4.5SHの機能解析をしようではないか!ということに相成りました。
さてここで4.5SHなのですが、このマイナーなncRNA。知る人ぞ知る、つまるところ知らない大部分の人は知らない、かなりオタクなncRNAなのですが、さすが日本のRNA業界。発生屋だった僕がGomafuをおっかなびっくりRNA関連ミーティングで発表した時にいろいろ質問してくださった富山大の広瀬さん(注:北大の廣瀬さんではない)が、なんとなんと、4.5SHの発見者である原田文夫先生門下ということで、色々お詳しいのです。これが。RNA業界日本、やはり奥深いです。色々教えていただき、4.5SHの研究の現状というのをおぼろげながらつかむことができました。
ここで簡単に広瀬さんに教えていただいた4.5SHのまとめです。
1) 4.5SHはマウスやラットではものすごく発現量が多いけれども、齧歯類、しかも齧歯類の一部の種でしか見られないncRNAである。
2) 4.5SHは細胞質にあるという報告もあるけれども、どう考えても核内ncRNAである(new)。
3) 4.5SHはレトロトランスポゾンSINE B1とよく似ているけれども、転移能はなく、ゲノム上にクラスターで存在している。
4) 4.5SHの機能は全然わかっていないし、ぜひ調べてくださいな。
5) 4.5SHのHは原田先生のイニシャルのHだという説もある。
4)に勇気付けられ、5)に大笑いし、2)はpracticalに役に立ち、ともあれもう4.5Shの虜になってしまって、いろいろやってみようと。餅は餅屋ではないですが、やはりその道の専門家とディスカッションしていると、自然にやるべきことが見えてきて、とりあえず方針ははっきりしました。つまり、
↓4.5SHはレトロトランスポゾンSINE B1とすごく似ている。
↓4.5SHはレトロトランスポゾンではないので、サイレンシングされておらず、発現量も多い。
↓4.5SHは核内に局在する。
↓SINE B1がアンチセンス方向に挿入されているmRNAはごまんとある。
↓以上を考えれば、4.5HSはSINE B1がアンチセンス方向に挿入されたmRNAとハイブリして、なにかしてる(たとえば核内に繋留して発現抑制とか)のでは??
なら、やること決まっています。SINE B1をセンス方向(コントロール)もしくはアンチセンス方向(実験区)にUTRに挿入したレポーターLuc遺伝子を作って、細胞に放り込んだらどうなるか?
予想は的中。UTRにアンチセンス方向にSINE B1を挿入したレポーターはセンス方向にSINE B1を挿入したレポーターに比べ、明らかに活性が低い!!しかも、レポーター遺伝子のmRNAの細胞内局在を、細胞質もしくは核分画からとってきたRNAで調べてみると、どうやらアンチセンス方向にSINE B1がレポーター遺伝子のUTRに挿入されると、4.5SHと相互作用して核内に繋留されるということがわかってきました。
レポーター遺伝子の挙動が分かったら、当然次にやるべきは内在のmRNAの挙動の解析です。mRNAのUTRにSINE B1がセンス方向に挿入されたmRNAと、アンチセンス方向に挿入されたmRNAとで、細胞内局在に変化が見られかどうか。たとえば、4.5SHノックダウンをしたら、それらのmRNAの細胞内局在が変化するのではないか。今だったらRNAseqとかするのでしょうが、とりあえずそんな技術もなかった当時僕らがしたのは、古典的なノザンブロット解析。それを実施してみると、SINE B1がアンチセンス方向に挿入されたmRNAは、4.5SHをノックダウンすると、細胞質に漏れ出てくるということが、スメアなバンドの総体としてではありますが、確認することができました!! ちなみに、これらのアイデア、すべて実験をしていた石田くんのアイデア。次々と思いどおりに進んで行く4.5SHプロジェクトは、まさに蜜の味でした。しかしこれがこの夢のような話のピークだったとは、、、(誰がおもいうかんだであろうか(いやない)(反語的表現))
ちょっとここらでさすがにつかれてきたので、コーヒーブレーク。。。次はほんとのほんとの最終章、キメの実験をしたらそれまでの仮説に合わないデータが出てきて、5年かけて集めたデータのほとんどが吹き飛んでしまったけど、却ってシンプルでわかりやすい論文に?の巻です。


 2020
2020