まずは、Pubmedで検索して、csvリストの出力をします。普通に検索して、チェックボックスにチェックを入れて、右上のSend toのボタンをクリック、Fileを選んで、FormatのプルダウンでCSVを選んで、Create Fileボタンを押すだけですね。
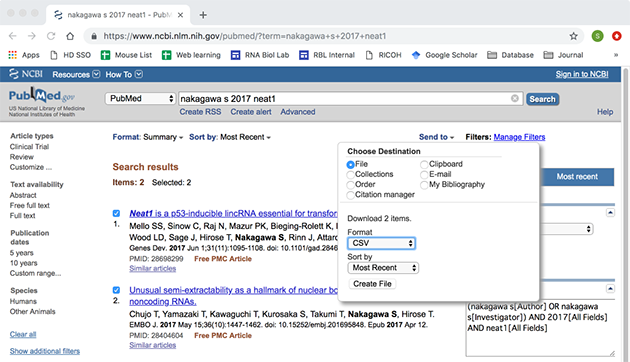
そうすると、ダウンロードフォルダにPubmed_result.csvというファイルが保存されます。Pubmedのデータフォーマットもあまり綺麗でなくて、全部の情報をカンマで区切ってくれていてくれれば良いのですが、4列目とかは雑誌名と発行年、ページ数からdoiまでごっちゃの情報になっています。そこで正規表現で必要な情報を取り出して、モンカ様のお気に入りフォーマットに整形します。科研費マニュアルによりますと、モンカスペシャルフォーマットは『DOI[160 桁], 著者名[160 桁], 論文標題[400 桁], 雑誌名[200桁], 巻[30 桁], 発行年[4 桁], 最初と最後の頁[15 桁], 査読の有無[1 桁], 国際の共著[1 桁], オープンアクセス[1 桁]』だそうなので、
setwd("~/Downloads")
library(stringr)
#Pubmed export file to KAKEN excel file
df <- read.csv("pubmed_result.csv", skip = 1, header = FALSE)
df["doi"] <- gsub("\\.$","",gsub("\\. .*$","",gsub("^.*doi: ","",df[,4])))
df["journal"] <- gsub("\\..+$", "", df[,5])
df["volume"] <- gsub("\\(.*$","",gsub("\\:.*$","", gsub("^.*;","",df[,4])))
df["year"] <- gsub("^.*\\. ","",df[,5])
df["page"] <- gsub("\\..+$","",gsub("^.*?\\:","",df[,4]))
df_KAKEN <- df[,c(13,3,1,14:17)]
df_KAKEN[c(8:10)] <- "" #dummy values
write.table(df_KAKEN, file = "KAKEN.csv",sep = ",", col.names = FALSE, row.names = FALSE)みたいな感じでしょうか。うーん。美しくない。実に美しくない。左側と右側の情報をトリミングする方法がわからなくて入れ子のgsubになってしまい、あまりにも美しくないスクリプトでRの師匠のY本君に怒られそうですが、まあ、目的が達せられれば良しとして。あとはダウンロードフォルダに生成されたKAKEN.csvファイルを科研費システムのcsvファイル取り込みボタンで取り込み、右の登録ボタンを押すだけです。査読の有無とか国際共著のフォーマットはわからなかったので、ダミーの空白データを入れているので、適宜チェックボタンを押せば完成!
このスクリプトが役にたつぐらい論文書くんだ。来年こそ。


 2020
2020