このプロジェクトがスタートしたのは今から6年ぐらい前まで遡るのですが、和光理研の眞貝さんや石井さんが中心となってエピゲノム関連のプロジェクトが立ち上がり、こりゃ面白そうだということで、細胞タイプ特異的なエピゲノム解析を開発する、というテーマでメンバーに加えていただきました。ちょうど10年前になりますが、2008年にTRAP法(タグつきのリボゾームを特定の細胞で発現させることで、FACS等を用いた細胞の単離という非生理的なステップを経ずに、インタクトな状態で細胞タイプ特異的な発現プロファイルを解析する手法)の論文が発表され、賢い方法もあるものだなあ、と感心していたのですが、当然同じようなことをタグつきヒストンでやって特定の細胞でのエピゲノム修飾を調べている人もいるだろう、ということでいろいろ文献を調べてみたところ、これが意外なことに、無いんですね。そもそも、ChIPによるエピゲノム解析は培養細胞を用いたものがほとんどで、組織レベルでやっている人となるとグンと数が減ってきます。ましては組織の中の特定の細胞で、となると、FACSを使って細胞を集めて、という例はいくつかあったのですが、組織そのままというのは皆無。それでも僕が知らないだけかもしれないなあ、ということで、これは専門家に聞くに如かず。えーと、特にターゲットにしたい神経系の組織丸ごとエピゲノム解析をしている人は、、、いない。いない。半径1キロ以内に知り合いでやっている人はいない。検索範囲を広げて、知り合いというレベルをこっちは知っているけれども向うは知らないとこまで下げて、あっ!後藤由紀子さんって神経系のエピゲノム解析やっておられるんだ!ということで、かねてよりファンだったこともあり押しかけディスカッションのお時間をいただいて相談に伺ったところ、うーん、面白いんじゃないですか、やっている人はいないと思いますよ、ということで、勇気百倍。Creの組換えが起きるとタグつきのヒストンを発現するマウスで、proof of principleとして大脳皮質のCamk2a陽性ニューロンあたりを使ってみれば良いと思いますよ、とのアドバイスもいただき、早速プロジェクトを始動することにしました。
何せ先立つものは人材、ということで急遽公募を出して当時横浜理研におられた水戸さんに参加してもらえることになり、キックオフミーティングで組織のChIPなら色々やっておられた神戸理研の工樂さんチームの門田さんにもバックアップしていただき、パイロット実験を色々開始したのですが、結局一番難しかったのは、バックグラウンドをどれだけ減らせるか、ChIPの効率をどれだけ上げられるか、というところでした。このやり方だと、一回FLAG抗体で目的の細胞で発現させたヒストンH2B-FLAGでChIPを行い、それを溶出して二回目のタンデムChIPを行うことになります。ステップが1ラウンド増えるのでS/N比と効率を上げないといけないわけですが、あれやこれや試してみてバックグラウンドを減らすのに一番効果があったのは、in situ hybridizationでよく使われているBlocking Reagent(要は精製カゼインですね)をブロッキング剤として使うこと、効率を上げるのに一番効果があったのは、これもin situ hybridizationでよく使われているデンハルトを加えること、でした。まあ、他のものでも代替は効くのでしょうが、今んとこうちのラボでの伝説は、ChIPバッファーにデンハルトとBlocking Reagentを加えましょうね、というものです。こうやってまたどうでも良いかもしれない伝説が増えていく。CanGetSignalを使ったらもっとうまくいくのかもしれませんが、試してません。一度うまく行ったプロトコールは変えないというのが実験屋の鉄則。。
実はこのプロジェクト、老化したマウスと若年マウスの神経細胞でエピゲノム修飾が変わっているのか否かを確かめるのが当初の目的でした。ですので、タグつきヒストンを発現させたマウスが老化するまでじっくり待ちましょう、ということでのんびり構えていたのですが、僕自身が北大に異動することになり、そんなにのんびりやっているわけにもいかなくなってきてしまいました。そもそも、僕の見通しが全くのところあまちゃんで、老化マウスn=5ぐらい用意しておけばなんとかなるよね、とか思っていたのですが、ぽちぽち飼育中にお亡くなりになっていくのですよね。そのほかさまざまな事情で、老化マウスのデータを取っている場合ではない、という状況になってしまい、とりあえず、生物学的に何か新しい発見があるかということは置いといて、細胞タイプ特異的にエピゲノム修飾を解析することができるんですよ、という論文としてこれまでの成果をまとめることになりました。
NGSを用いた解析でよく言われるのは、データ解析の部分が重要だということ。これからの時代ベンチ屋でもある程度の解析ができないといけない、というのは常に思っていることなのですが、なかなか、これが、難しいというのは間違いありません。マッピングしてIGVで見てみて、MACSでピークコールして、ぐらいであればなんとかなるのですが、というか水戸さんがご自身でどんどん解析を進めてくださっていたのですが、その一歩先、というところになると、なかなかベンチ屋では手に負えないところがあります。うーむ、、、と、困っていたところ、思いもかけぬ救世主登場。理研のテクニカルスタッフを北大に連れて行くわけにはいかないので、水戸さんの再就職先をいろいろ探してもらっていたところ、ちょうどタイミングよく新しく理研に着任される岩崎さんのところに移籍できることが早々に決定。しかも、岩崎さんは留学先でのribosome profilingのお仕事で、データ解析はお手の物。ん?岩崎さんって、昔、泊さんのところでバキバキの生化学をやっていたベンチ屋だよね。。。出来る人は出来る。それと比べて私は、といじけていては何も始まらないので、ひたすらダイセクションはできる、ということに徹して、チームワークでなんとか一通りのデータを揃えることができました。
この実験でわかったことは、タグつきヒストンを特定の細胞で発現させることで、細胞の単離というステップを経ずに、細胞タイプ特異的なエピゲノム修飾を調べることができるということ。これはある意味予測されていたことで、驚きはあまりありません。良かったねえー、でおしまいなのですが、意外だったのは、岩崎さんがいろいろ公共データべースとの比較を行って、僕らが調べたH3K4me3のプロモーター領域の修飾と、その遺伝子の転写産物の発現量をリボゾームIPで調べた結果が、あまりにも違うらしい、ということでした。
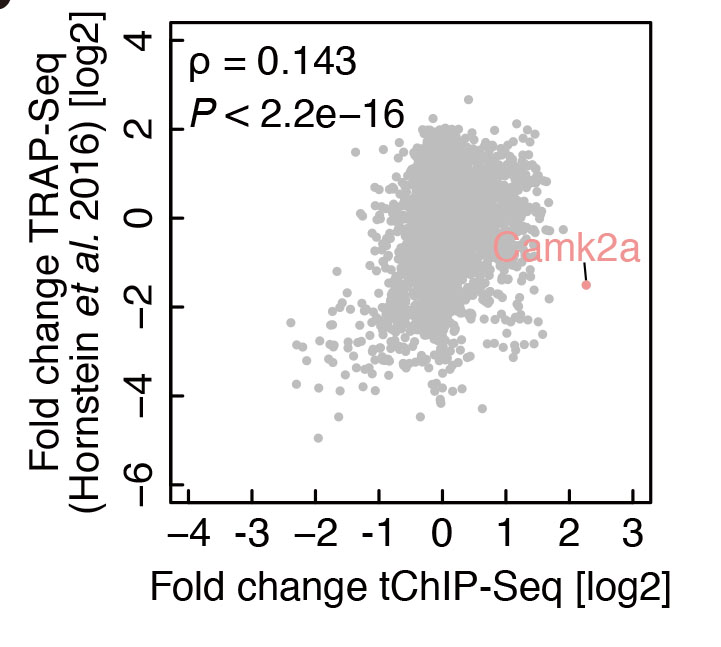
H3K4me3の修飾は、例外はいろいろあるにせよ、大まかに言えばその遺伝子の発現量を反映しています。ところが、先に触れたTRAP法、すなわち、僕らと同じCamk2a陽性細胞に発現させたタグつきリボゾームと相互作用しているRNAの結果が、あまりにも一致しない。ほとんど相関なし。これって、僕らのメソッドがアカンタレということなのかなあとちょっと冷や汗たらりだったのですが、岩崎さんの渾身の解析で、樹状突起やシナプスなどに濃縮されていることが知られている遺伝子がTRAP-seqではunder-representedされているということがわかってきました。よくよく考えてみるとそれは非常にreasonableで、局所輸送されているRNAはRNA granuleの中に入っていて、積極的に翻訳されているわけではありません。ですので、神経細胞のように複雑な形態をもち遠位に輸送されるRNAを数多く持つ場合は、プロモーターのH3K4me3の修飾を調べたほうが遺伝子発現プロファイルをより正確に調べることができるのかもしれません。これは全く予想外の結果で、公共データベースを積極的に利用することの大切さをしみじみ感じました。
この細胞タイプ特異的エピゲノム解析の手法を、僕らはtChIP(tandem ChIP)と名付けたのですが、必要な材料(体全体でヒストンH2Bを発現するマウスとCre依存的にタグつきヒストンH2Bを発現するマウス)は理研バイオリソースセンターから供給されています。特にライセンスは設定していないので(理研とのMTAは必要ですが)、ご興味のある方はお気軽にお使いください。組織全体の1割を占める細胞タイプであれば、かなり正確にその細胞のエピゲノム状態を調べられるはずです。それよりも存在比が少ない場合(全体の1%以下とか)はノイズに埋もれてしまって解析できない可能性が高く、何らかの技術的改変が必要であると思います。
このプロジェクト、開始から6年も経ってしまったので、少々古臭い技術になってしまいました。この間、single cell RNA seqとか、small scale ChIPでの技術革新は目覚ましく、実際、最初に投稿した雑誌では、「で、何か?」みたいな扱いでした。その批判は実際そうだと思いますし、素直に受け止めるとして、XXseqと名のつく手法をきちんと発表することができたのは、まさに望外の喜びでありました。老化マウスでの解析ができなかったのが心残りではありますが。
ちなみに、この論文のデータ、もちろんncRNA研究の足しにもなります。神経細胞で発現しているncRNAのプロモーター、例えばGomafuちゃんのプロモーターには、脳全体の組織でのデータに比べて、はっきりとしたピークがみられます。
.jpg)
tdfファイルは手軽にダウンロードできるところに置いてありますので、暇なときにでもちょろっと見ていただけると幸いです。


 2020
2020