次にターミナルに移動してルンルン気分でコマンドラインを入力します。あらかじめwndchrmフォルダに移動しておくと、出力されたファイルを探しに行かなくて楽です。
cd /Users/Nakagawa_imac/Documents/wndchrm
あとはwndchrmのコマンドを打ち込むだけ。まずはwndchrm trainコマンドで、親フォルダの場所を指定して、画像の各特徴をまとめたファイル.fitを作らせます。ここではwndchrm_demo.fitにしてみましょう。-mはマルチコアを使うためのオプション。-lはなるべくたくさんのカテゴリーについて特徴値を計算するためのオプションです。カラーのときは-cのオプションをつけるようです。
wndchrm train -m -l wndchrm_demo wndchrm_demo.fit
すると、画像が置いてあったフォルダに.sigというファイルが生成されていきます!
次々とスクロールするターミナルのこういう画面を見ているとなんか働いてる気がする。知らない人が横から見たら、データサイエンティストに見えるかしらん。
.sigファイルをテキストエディットなどで開いてみると各カテゴリーにおける特徴値を見ることができます。Chebyshev-Fourier CoefficientsやらOtsu Object FeaturesやらHaralick Texturesやらありますが、何のことやらさっぱりわかりませんね。まあ、コンピューター将棋で言うところの、<玉ー銀ー金>や<玉ー馬ー金>といった三駒関係みたいなものでしょう(余計わからない)。ともあれ、画像を特徴づける2919項目について数値化しているようです。我々が発表論文数、招待講演数、H-indexなどで評価されるようなものでしょうか(わかりやすい)。終了するとwndchrmフォルダのところにwnchrm_demo.fitというファイルが生成されてきます。
次に、これらの評価値でどれぐらい上手に分類できるかテストさせます。何も指定しないと、全体の3/4の画像を無作為に抜き取って評価関数を作り、残りの1/4の画像でどれぐらいの正確さで分類できるか計算します。このテストを30回実行させて、どれだけ正しく分類できたかの結果をwndcrhrm_demo_result.htmlとして出力させてみます。
wndchrm test -m -l -n30 wndchrm_demo.fit wndchrm_demo_result.html
さて、結果ですが、htmlファイルを開いてみます。たとえば、
| A_Cell | B_Cell | C_Cell | D_Cell | Total Tested | Per-Class Accuracy | ||
|---|---|---|---|---|---|---|---|
| A_Cell | 143 | 7 | 0 | 0 | 150 | 95.3 +/- 3.4% | |
| B_Cell | 2 | 148 | 0 | 0 | 150 | 98.7 +/- 1.8% | |
| C_Cell | 0 | 0 | 83 | 67 | 150 | 55.3 +/- 8.0% | |
| D_Cell | 0 | 0 | 64 | 86 | 150 | 57.3 +/- 7.9% |
Intervals based on 95% confidence using Normal Approximation method.
| A_Cell | B_Cell | C_Cell | D_Cell | |
|---|---|---|---|---|
| A_Cell | 1.00 | 0.05 | 0.00 | 0.00 |
| B_Cell | 0.03 | 1.00 | 0.00 | 0.00 |
| C_Cell | 0.00 | 0.00 | 1.00 | 0.90 |
| D_Cell | 0.00 | 0.00 | 0.87 | 1.00 |
| A_Cell | B_Cell | C_Cell | D_Cell | |
|---|---|---|---|---|
| A_Cell | 0.9599 +/- 0.0242 | 0.0395 +/- 0.0242 | 0.0002 +/- 0.0001 | 0.0004 +/- 0.0003 |
| B_Cell | 0.0247 +/- 0.0173 | 0.9753 +/- 0.0173 | 0.0000 +/- 0.0000 | 0.0000 +/- 0.0000 |
| C_Cell | 0.0000 +/- 0.0000 | 0.0000 +/- 0.0000 | 0.5475 +/- 0.0417 | 0.4524 +/- 0.0417 |
| D_Cell | 0.0001 +/- 0.0000 | 0.0000 +/- 0.0000 | 0.4534 +/- 0.0354 | 0.5466 +/- 0.0354 |
95% confidence intervals
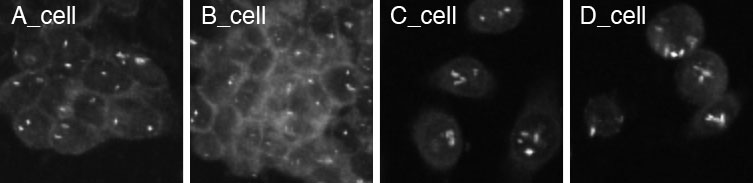
Confusion Matrixという項目。これはA_cellのフォルダに入っていた画像をランダムに5つ選んで30回テストしたときに、それがA_Cellとして分類されたのが143回、B_Cellとして分類されたのが7回、CやDと見間違えることはなかった、ということになります。ですので、Per-Class Accuracyは95.3%。かなり正確に分類できています。一方、C_Cellの場合は67回もD_Cellとして分類されてしまっています。ほぼ半分も間違えてるんかいな?wndchrm使えんなあ、、ではなくて、実はこれ、CとDは同じ細胞なんです。ですので、正しく働いているということになります。こんな人を試すような陰湿なことをしても怒らないし惑わされないのがコンピュータの偉いところですね。はい。
また、前回インストールしたphylipの場所を-pオプションに続けてスペースを入れずに指定すると、それぞれの細胞がどれぐらい似ているのかというのを系統樹を書いてくれ、ファイル名.psの形式で出力してくれます。たとえば僕の場合phylipはこんな感じ
のフォルダ構造で入っていますので、
wndchrm test -m -l -n30 -p/usr/local/Cellar/phylip-3.695 wndchrm_demo.fit wndchrm_demo_result.html
になります。で、出てきた結果がこちら。
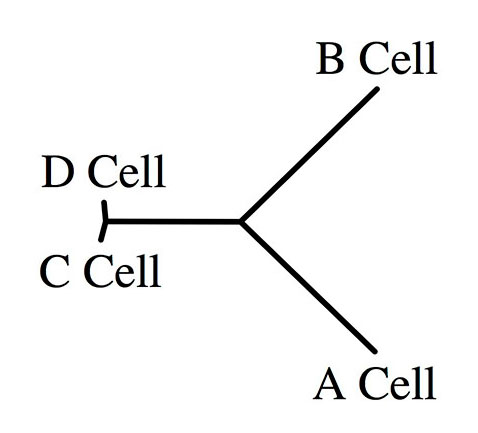
CとDはほとんど変わらんということがきちんと見えています。
ついでに、未知の細胞の画像をwndchrmに投げて、どのカテゴリーに分類できるか判断させることもできます。たとえば、あんまり意味ないですがデモ画像のA_Cellの#1の画像を判断させるときは、
wndchrm classify -m -l wndchrm_demo.fit /Users/Nakagawa_imac/Documents/wndchrm/wndchrm_demo/A_Cell/A_Cell_#1.tif
結果はターミナルのウインドウに出てきます。当然ながらこの場合、Aに分類されます。
以上、wndchrmはインストールも使うのも非常に簡単、中身はわからないけれどもかなり正確、冷静に客観的に画像の類似度を判断してくれるツールとして、かなり優秀なのではないかと思っています。ノックダウンの効果にイマイチ自信が持てないときなど、画像を撮りまくってwndchrmに投げて、分類精度が80%を超えてくるようであればまず効果があるとみて間違いなし、と自信を持てるのではないでしょうか。
あと、熊大の斉藤さんから教わったポイントですが、
画像のサイズと枚数は各条件で揃えておいたほうが良い。 画像は多ければ多いほど良いが、20-30枚あればかなり精度の良い分類ができる。 当然ながら同じ条件(露出)で撮影しないと結果がばらつく。
僕が学んだのは、相変わらずコンピュータ音痴ぶりを発揮していますが、
ファイル名にはスペースを入れない(間を空けたいときはアンダーバー_を使う)。 -nや-pなどのオプションの後にスペースを入れない(入力フォルダとして認識されてしまう)。 現時点ではphylipsはhomebrewでインストールしない。 マニュアルをちゃんと読む。 困ったらググる。 それでも解決しない時は斉藤さんに聞く。
というわけで、サルでも使えるwndchrmの紹介でした。実験医学の単行本に斉藤さんのはるかに丁寧な解説がありますので、羊土社の回し者ではありませんが、これを機会にぜひこちらの本も。


 2020
2020