
ここ数年で、コテコテのベンチ屋でもある程度の次世代シークエンサーのデータ解析ができる環境がどんどん整ってきました。なんのセットアップも必要なくNCBIのBLAST感覚で手軽に利用できるのがクラウドベースの解析環境で、老舗のGalaxyに加え、最近はセルイノベーションプログラムで整備された遺伝研のMaserなどがきめ細やかなサービスを提供してくれています。さすが遺伝研。また、とっても素人friendlyな「次世代シークエンサーDRY解析教本」などのスグレモノ書籍も出版され、my Macに各種ツールをインストールして自前でなんちゃってNGS解析しているwetな学生さん(性格がwetなわけではない)も数多くおられると思います。そう。もう、仕事をDryとWetなんて分ける時代ではない。今や時代は、理研CDBの工樂さんがおっしゃるところの、"Moisture"で行こう!です。(2/8追記:ったくこれだから素人は、、、というミスがありましたので訂正入れました)
「ネット」=「妖しいもの」というのが一昔前は定番だったと思いますが、今ではすっかり市民権を得て、新聞やテレビよりも信頼できる、なんて言っている人もいるようです。その真偽はともかくとして、ネット上のSNSは情報収集という観点からはなかなかバカにならないものがありまして、先日の当ブログのエントリーにも、早速rnacintosh LC475さんからツッコミがありました。
なるほど!
— rnacintosh LC475 (@68040LC) 2016年12月16日
僕ならファイルをfastaに書き換えて
cat Arraystar_probe_list.txt | sed "s/^/>/g" | tr ',' '\n' > out.fa
んでもってbowtie https://t.co/BPWXan0170
ちょっと前に谷上さんも書いておられましたが、今の時代、ちょいと調べてみようか、と思った実験の結果が公共データーベースにのっているというのはよくある話で、自分の実験を組み立てる前にデーターベースを当たってみて情報収集をするというのは、ムダを省く上でも必須の作業になりつつあります。個人的に暇な時にちょいちょい見ているのがNCBIのGEO Profilesで、my favorite geneの名前を入れるだけで、その遺伝子が変動しているような解析をずらりと並べてくれます。気になる解析があればNCBIのWebインターフェースからちょっとした解析するも良し。GEO datasetから生データーを落としてきて自前で解析するも良し。何せ便利な世の中になったものです。
ラボで行われている実験の伝統や伝説のほとんどはただの迷信、というのは良くある話で、プラスミドプレップする時のリゾチームだとか、コンピテントセルのヒートショックだとか、RNA実験に使う水のDEPCだとか、まあ少しは変わるのかもしれないけれども本質的には変わらないよね、という話はゴロゴロしています。FISHにおいても伝説がありまして、それは、
DIGラベルしたプローブはマウスのモノクロでしか検出できない。
です。したがって、マウス由来の抗体で何かのマーカータンパク質を染めて二重染色したいときは、FITCラベルしたプローブをウサギのポリクロで見ないといけない、これはDIGラベルより若干感度が落ちるから困ったねえ、というのが勝手に僕が作った伝説ですが、今回、総括斑の超解像顕微鏡用の予算に少し余裕があったので、この伝説を検証することにしました。
結論からいくと
伝説はやっぱりただの伝説。DIGのポリクロめちゃそこそこ使える、でありました!!!。
2016/4/22追記:いろいろ試してみると、現状どうもやはりそこそこ使える、のレベルで、FITC/anti-FITC(Rb)/anti-Rb Cy2の組み合わせにはかなわないようです。難しいものです。
京都大学ウイルス研究所 大野研究室に所属する研究員の竹岩と申します。
2015年9月9日〜11日に開催されたin situ / 超解像顕微鏡講習会にて中川先生にご教授
いただきましたin situサンプル調整の方法(秘技)につきまして、後半2日目のプロトコールを
まとめましたので投稿いたします。
2015年9月9日〜11日に理研(和光)と東大分生研で開催されたin situ / 超解像顕微鏡講習会において、中川研で教わったin situサンプル調整の秘技(1日目のプロトコール)をまとめました。
超解像であろうと通常のコンフォーカルであろうと、はたまた普通の蛍光顕微鏡であろうと、二重染色の解釈には定量解析がしばしば要求されます。共局在の定量解析の定番といえば、やはりフリーソフトImageJ。ポケモンではないですが、ImageJには進化型があるらしく、その名もFiji(Fiji Is Just ImageJ)。早速こちらのページからインストールすると、PluginやAnalyzeプルダウンメニューにわんさかアイテムが入っていて、お得感満載。共局在の定量化はAnalyze>Colocalization>Coloc 2を使うようです。
インストールが済んだところで、いよいよwndchrmを使ってみます。手始めにこの細胞
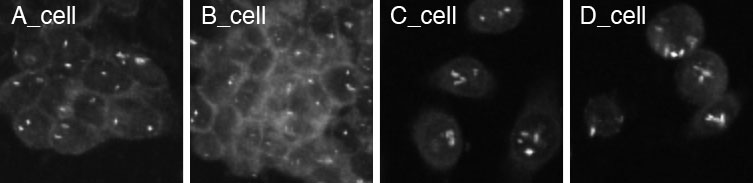
たちを分類出来るか見てみましょう。まずはこの記事の下のほうにある「添付ファイル」のところにあるwndchrm_demo/zipをダウンロードして解凍してみてください(こちらからでもダウンロードできます)。wndchrm_demoというフォルダの中に、細胞4種類A, B, C, Dそれぞれについて20枚の画像が別々のフォルダに入っています。ここではDocumentフォルダ(「書類」フォルダ)直下にwndchrmというフォルダを作って、解凍した親フォルダのwndchrm_demoをその中に入れた時の操作例を示します。ちなみに画像は基本的にTIFF形式。白黒でもカラーでも使えます。今回は白黒です。
タイトル通り、現在僕のラボで使っているin situ関連試薬のカタログ番号のまとめです。随時更新、追加していきますが、こっちのほうが良いとか、これはアカンとかありましたら、コメントいただけると助かります。
ミニプレップばりに基本中の基本で、経験ある人にとっては常識すぎて今更感漂う手順ですが、初めての人にはそれなりに緊張感漂うRNAプローブの作り方をまとめておきます。